DNA甲基化与癌症之间不得不说的“秘密”
DNA甲基化是哺乳动物中研究最为深入的表观遗传修饰之一。在正常细胞中,DNA甲基化可以有效地调控基因表达水平,某些抑癌基因的失活是由于启动子区域的高甲基化导致的。大量研究表明,在大多癌种中DNA甲基化可导致大范围的基因沉默。除启动子区域和DNA重复序列中的甲基化水平改变外,甲基化还与非编码RNA(如肿瘤抑制作用相关的microRNA)的表达调控有关。DNA甲基化水平与肿瘤发生发展过程的联系,鼓励着我们不断的解码人类表观基因组。本篇将由小编带大家了解DNA甲基化与癌症之间的关系。
什么是DNA甲基化?
哺乳动物细胞中的DNA甲基化是在DNA甲基转移酶(DNMT)的催化作用下,在胞嘧啶碱基(5-甲基胞嘧啶;5mC)的碳-5位添加甲基,通常发生在胞嘧啶-鸟嘌呤二核苷酸(CpG)中。在人类基因组中,约有2800万个CpG位点,且这些CpG位点的分布并不均匀,而在基因组的某些区域,CpG保持或高于正常概率。CpG岛主要位于基因的启动子和第一外显子区域,其功能是通过甲基化与去甲基化调控下游基因的表达。如果CpG岛发生高甲基化,基因表达就会被抑制。大多数CpG岛的长度为500-1000个碱基对(bp),与大部分基因组不同的是位于CpG岛内的CpG位点通常在正常体细胞中为未甲基化状态。
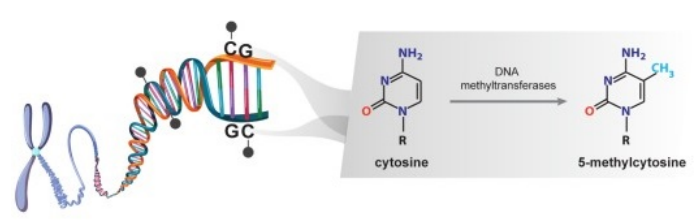
图1 DNA甲基化原理(Skvortsova K et al.,2019[1])
参与该过程的DNA甲基转移酶包括DNMT1,DNMT3A及DNMT3B。DNMT1酶可识别半甲基化的DNA,并保持现有甲基化模式;DNMT3A和DNMT3B则在未甲基化的胞嘧啶上添加甲基基团。除5-甲基胞嘧啶外,5-羟甲基胞嘧啶(5hmc)作为胞嘧啶的甲基化中间产物被一同认定为两种最常见的表观遗传标记。通过双加氧酶家族TET蛋白,将5mC氧化为5hmc[2],而DNA的5mC和5hmc水平在肿瘤发生发展中发挥了重要的作用。
甲基化与癌症
DNA甲基化对于维持细胞正常功能及发育至关重要,肿瘤细胞中甲基化特征与正常细胞大有不同,在癌细胞中可以同时检测到低甲基化和高甲基化水平的改变。其实,在肿瘤发生发展的过程中,表观水平就已经出现了异常,DNA甲基化整体上发生改变。一般而言,CpG区域的甲基化水平总体下降,可导致基因组的不稳定性,但较少地激活沉默的原癌基因。
众所周知,癌症的发生、发展伴随着DNA甲基化模式的改变,包括了逆转录元件、着丝粒和原癌基因的DNA低甲基化,以及与基因抑制相关的关键基因调控元件(如远端增强子和启动子转录起始的重叠区域)的甲基化。如图2所示,在整个基因组的所有基因调控元件,正常细胞与癌症细胞之间的DNA甲基化模式存在广泛差异。正常基因组中大部分CpG位点都携带着5mC,而远端增强子元件及CpG岛区域对甲基转移酶DNMT的活性具有抗性。癌细胞主要表现特征为整体范围的失去甲基化遗传学修饰,反而增强子和启动子区域内出现异常的甲基化位点。这种甲基化分布的改变,导致肿瘤抑癌基因的表达受抑制,并伴随着原癌基因表达的增加,从而进一步推动了肿瘤的发生、发展。
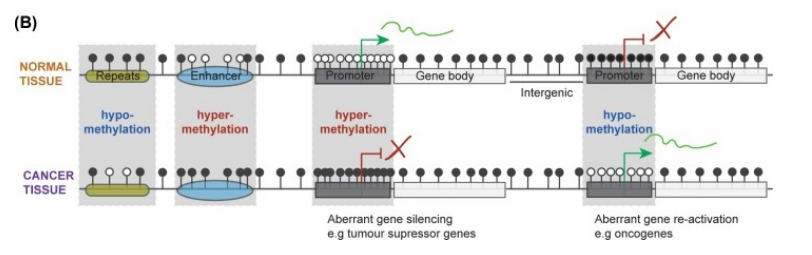
图2 正常细胞与癌细胞DNA甲基化模式的差异
(Skvortsova K et al.,2019[1])
DNA甲基化将成为常见肿瘤标志物
鉴于正常细胞与癌细胞的DNA甲基化模式存在有显著差别,使得DNA甲基化有成为筛查指标的可能。因此,DNA甲基化的生物标志物必须克服当前的局限性并且优于现有诊疗手段的敏感性及特异性或通过微创方法具有更好的可接受性。大量研究表明,在癌症诊断中DNA甲基化在预测患者的预后、指导治疗方案的选择、监测治疗效果等方面有显著提升。
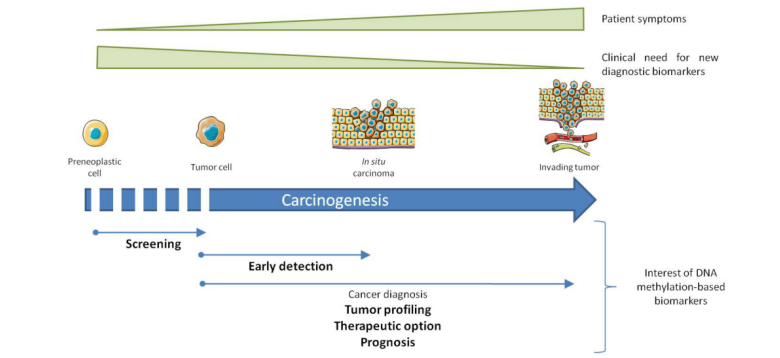
图3 DNA甲基化标志物在癌症诊断中的意义
(Delpu, Y et al.,2013[3])
癌症早期诊断
早期癌症的诊断对于患者的治疗和预后都非常重要。McCluskey等研究显示p16基因高甲基化差异可区分良性和恶性卵巢肿瘤,并且远端启动子在33%的低度恶性潜在肿瘤中被甲基化,而在癌中为5% [4]。同样,又一研究表明编码miR-148a的基因的甲基化水平是鉴别胰腺癌和慢性胰腺炎的潜在诊断工具[5]。
癌症分型
DNA甲基化可以用于癌症分型。不同类型的癌症之间存在DNA甲基化模式的差异,因此DNA 甲基化模式可以作为改进肿瘤分类的有力工具。如在49名急性淋巴细胞白血病(ALL)患者中使用基于高分辨率DNA甲基化的生物标志物(67,487个探针)可以区分不同的ALL亚型并影响临床结果[6]。
癌症预后评估
除早期诊断和癌症分型之外,DNA甲基化还能预测肿瘤对治疗的反应并改善患者的预后。例如,40% 的神经胶质瘤患者中MGMT启动子的DNA高甲基化与肿瘤对基于烷化剂的常规化疗的耐药性直接相关。此外,正常细胞在肿瘤中的积累会影响对肿瘤中MGMT表达的评估。更重要的是,它与肿瘤消退和延长的总生存期和无病生存期相关[7]。
.癌症治疗监测
DNA甲基化可以用来监测癌症治疗的效果,例如化疗、放疗等,根据DNA甲基化水平的变化来评估治疗效果。对于淋巴瘤患者,治疗后DNA甲基化水平的变化可以用来评估治疗效果。
DNA甲基化的优势
1
高度的组织特异性
表观遗传的变化通常发生在肿瘤早期,并具有组织和癌症类型特异性。不同组织和细胞类型的DNA甲基化模式不同,可用于确定肿瘤的类型和来源。因此,相关基因的甲基化状态是肿瘤发生的早期敏感指标,可认为是一种极具前景的肿瘤标志物。
2
高度的稳定性
相比其他生物标志物,如蛋白质和RNA,DNA甲基化在样本采集、运输和贮存过程中更加稳定,可以更好地保持其完整性和可靠性。
3
高度的灵敏性
DNA甲基化检测可以在非常早期的肿瘤发展阶段进行检测,甚至在肿瘤形成之前就能够发现异常的DNA甲基化模式。
4
高度的准确性
DNA甲基化检测具有高度的准确性和可重复性,可以用于肿瘤的早期诊断、分型、分级和预后等方面。
5
高度的多样性
DNA甲基化检测可以应用于各种样本类型,包括血液、尿液、组织切片和体液等,具有广泛的应用前景。
几种特定的DNA片段甲基化检测方法
甲基化特异性PCR (MS-PCR)
此法经济实惠,无需特殊仪器,目前应用最广。主要是先进行甲基转化,随后进行引物特异性的PCR。MS-PCR中需要设计2对引物,检测MSP扩增产物,根据处理后甲基化或非甲基化DNA链的引物能否扩增出片段判断被测位点是否存在甲基化。
荧光定量法(Methylight)
其原理是使用荧光水解探针,在MSP扩增同时检测荧光强度,使定量检测甲基化成为可能。用亚硫酸氢盐处理DNA片段,并设计一个能与待测位点互补的探针,开展实时定量PCR。其优势在于高通量、高敏感性,可做到多样本、多基因位点的快速分析。但该法费用较高。
甲基化敏感性限制性内切酶PCR (MS-RE)
此方法利用甲基化敏感性限制性内切酶对甲基化区不切割的特性,将DNA消化为不同大小的片段后再进行分析。这种方法成本较低,甲基化位点明确,结果易判读,但是该方法样本需求量大,不适用于混合样本,酶若不完全消化可能引起假阳性。
甲基化敏感性变性梯度凝胶电泳(MS-DGGE)
其原理是DNA片段在变性梯度聚丙烯酰胺凝胶电泳时,凝胶中变性剂浓度自上而下呈梯度递增,当DNA片段到达与解链区域的T(解链温度)值相当的某一浓度位置时,DNA解链变为分枝状,移动减慢,停留在凝胶的的某一位置,则不同的DNA段被分离。此法不需预先知道CpG位点及样本序列,需要的样品量少,但是解链温度和DGGE的变性浓度梯度需要摸索,且容易漏检。
肿瘤早期筛查是降低恶性肿瘤死亡率的有效手段,是未来检测技术的发展方向。DNA甲基化检测具有提高早期肿瘤检出率、取样简单、无创便捷、灵敏度及特异性高等优势,可以有效降低我国恶性肿瘤发生率及死亡率,由此可见,DNA甲基化在肿瘤领域应用前景将十分广阔!
参 考 文 献
1.Skvortsova K, et al. The DNA methylation landscape in cancer. Essays Biochem. 2019 Dec 20;63(6):797-811.
2.Giorgia Gurioli. Epigenetic Characterization of Cell-Free DNA. Cell-free DNA as Diagnostic Markers pp 129-135.
3.Delpu, Y.; Cordelier, P.; Cho, W.C.; Torrisani, J. DNA Methylation and Cancer Diagnosis. Int. J. Mol. Sci. 2013, 14, 15029-15058.
4.McCluskey, L.L.; Chen, C.; Delgadillo, E.; Felix, J.C.; Muderspach, L.I.; Dubeau, L. Differences inp16Gene methylation and expression in benign and malignant ovarian tumors. Gynecol. Oncol 1999, 72, 87–92.
5.Hanoun, N.; Delpu, Y.; Suriawinata, A.A.; Bournet, B.; Bureau, C.; Selves, J.; Tsongalis, G.J.; Dufresne, M.; Buscail, L.; Cordelier, P.; et al. The silencing of microRNA 148a production by DNA hypermethylation is an early event in pancreatic carcinogenesis. Clin. Chem 2010, 56, 1107–1118.
6.Stumpel, D.J.P.M.; Schneider, P.; van Roon, E.H.J.; Boer, J.M.; de Lorenzo, P.; Valsecchi, M.G.; de Menezes, R.X.; Pieters, R.; Stam, R.W. Specific promoter methylation identifies different subgroups of MLL-rearranged infant acute lymphoblastic leukemia, influences clinical outcome, and provides therapeutic options. Blood 2009, 114, 5490–5498.
7.Esteller, M.; Garcia-Foncillas, J.; Andion, E.; Goodman, S.N.; Hidalgo, O.F.; Vanaclocha, V.; Baylin, S.B.; Herman, J.G. Inactivation of the DNA-repair gene MGMT and the clinical response of gliomas to

